Оргинальная статья
ТАКСОНОМИЧЕСКОЕ РАЗНООБРАЗИЕ БАКТЕРИАЛЬНОГО АНСАМБЛЯ В ЭМБРИОЗЕМЕ САМОЗАРАСТАЮЩЕГО ЗОЛООТВАЛА
Н.Б. Наумова1
 , И.П. Беланов1
, И.П. Беланов1
 , Т.Ю. Аликина2
, Т.Ю. Аликина2
Адрес: 1ФГБУН Институт почвоведения и агрохимии СО РАН, проспект Академика Лаврентьева, 8/2, г. Новосибирск, 630090, Россия
2ФГБУН Институт химической и фундаментальной медицины СО РАН, проспект академика Лаврентьева, 8/2, г. Новосибирск, 630090, Россия
Цель исследования: Целью исследования была оценка структуры и состава бактериальных ансамблей по разнообразию последовательностей генов 16S рРНК в молодой почве, формирующейся при самозарастании золоотвала.
Место и время проведения. В качестве объекта исследования был выбран эмбриозем самозарастающего в течение девяти лет золоотвала ТЭЦ-5 г.Новосибирска (54°59' с.ш., 83°03' в.д.), а в качестве фоновой почвы – серая почва под ненарушенным березовым лесом недалеко от золоотвала (55°00' с.ш., 83°04' в.д.); образцы почв отобраны из слоя 0-5 см в конце июля 2017 г. в трех индивидуальных повторностях.
Методология. Экстрагированную из почв тотальную ДНК использовали в качестве матрицы для амплификации вариабельного района V3-V4 генов 16S рРНК, и полученные ампликоны секвенировали на платформе Illumina MiSeq. Статистическую обработку данных проводили методами описательной статистики и анализа главных компонент.
Основные результаты. Всего в изученных образцах почвы было выявлено 4887 различных ОТЕ, относящихся к 306 родам, 212 семействам, 123 порядкам, 84 классам и 32 типам бактерий. На уровне типа основные доминанты бактериальных ансамблей эмбриозема и серой почвы были одинаковы (Proteobacteria, Acidobacteria и Actinobacteria), и по общему очертанию относительного обилия типов изученные бактериальные ансамбли были довольно схожи, что в целом свидетельствует о направленности почвообразования к серой почве. Большее обилие представителей типа Chloroflexi (среди которых есть аэробные и анаэробные термофилы, аноксигенные фототрофы и организмы, использующие органические галиды в качестве субстратов для анаэробного дыхания) в молодом эмбриоземе самозарастающего золоотвала согласуется с неблагоприятными условиями (резкие колебания температуры, высокие влажность, pH, содержание металлов) в этом педогенном субстрате на ранней стадии формирования почвы. Относительное обилие представителей порядков Rhizobiales, Actinomycetales и неклассифицированных представителей Acidobacteria_Gp6 и Acidobacteria_Gp3 было в 1,5-2,4 раза больше в контрольной почве, при этом доминируя и в эмбриоземе. Индексы α-биоразнообразия, характеризующие видовое богатство в каждой экосистеме (Као-1, Маргалефа, Фишера, число ОТЕ) были выше в серой почве, а выравненность и равномерность распределения последовательностей по различным ОТЕ, наоборот, были выше в эмбриоземе. Индекс Шеннона был одинаково высок (6,3) в обеих почвах.
Заключение. За девять лет самозарастания золоотвала в формирующемся эмбриоземе развился богатый видами выровненный по структуре бактериальный ансамбль, который уже на этом раннем этапе педогенеза на уровне типа был близок к таковому в серой почве под ненарушенным лесом, расположенном по соседству с золоотвалом. В бактериальном ансамбле молодого эмбриозема доминирует специфичный комплекс бактерий (Chloroflexi, Xanthomonadales, Geobacter, Aciditerrimonas, Iamiaceae и др.), осуществляющих выветривание педогенного субстрата. Присутствие нитрификаторов (Nitrospira), денитрификаторов (Reyranella) на фоне доминирования азотфиксаторов (Bradyrhizobium, Rhizobiales) свидетельствует о становлении всего комплекса процессов трансформации азота уже на ранних стадиях почвообразования в ходе спонтанного самозарастания золоотвала. Доминирование в формирующемся эмбриоземе бактерий, информация о последовательностях генов 16S рРНК которых отсутствует в соответствующих базах данных, подчеркивает необходимость более детального исследования почвенных микробных ансамблей, развивающихся на отвалах, в связи с большой площадью территорий, занятых такого рода техногенными объектами.
Ключевые слова:
: эмбриозем; серая почва; золоотвал; отходы ТЭЦ; бактериальный ансамбль; гены 16S рРНК; Illumina MiSeq; березовый лес; Западная СибирьЦитирование: Наумова Н.Б., Беланов И.П., Аликина Т.Ю. Таксономическое разнообразие бактериального ансамбля в эмбриоземе самозарастающего золоотвала // Почвы и окружающая среда. 2019. Том 2. № 3. e84. 10.31251/pos.v2i3.84
Введение
Ежегодно российская энергетика наряду с электроэнергией и теплом производит около 25 млн тонн золошлаковых отходов (Энергетика, 2019, основная масса которых годами хранится в золоотвалах. В настоящее время накопленный объем отходов теплоэнергетики в России достигает порядка 1,5 млрд тонн, а площадь золоотвалов превышает 20 тыс. гектаров (Шилов, Шилова, 2017). В Новосибирской области ежегодно от сжигания угля образуется около 800 тыс. тонн золошлаковых отходов, отвалы с которыми занимают более 1000 га (Калачев, 2007).
По окончанию эксплуатации золоотвалы требуют проведения работ по консервации, но в перспективе необходимо восстанавливать экосистемные функции и сервисы, в частности, почвенно-экологические функции поверхностного слоя отработанных золоотвалов как основного интерфейса взаимодействия всех компонентов экосистемы. Несмотря на довольно длительную историю изучения таких территорий, нельзя не согласиться с выводом о том, что многие физические, химические и биологические аспекты особенностей формирования и развития эмбриоземов на золоотвалах изучены недостаточно (Haynes, 2009).
Одним из наиболее экономически и технически эффективных способов консервации является стимулирование процессов самозарастания постиндустриальных ландшафтов. Почвенные микроорганизмы на начальных стадиях самовосстановления экосистем играют исключительно важную роль не только как агенты выветривания зольного субстрата и разложения органического вещества, но являются основной частью органического вещества, с которой собственно и запускается весь комплекс процессов его трансформации и гумификации в минеральном субстрате золоотвала. Однако почвосубстраты техногенных экосистем в плане инвентаризации биоразнообразия микробных сообществ современными метагеномными методами практически не изучены.
Целью исследования была оценка структуры и состава бактериальных ансамблей по разнообразию последовательностей генов 16S рРНК в молодой почве, формирующейся при самозарастании золоотвала.
Материалы и методы
Объекты исследования. В качестве объекта исследования для оценки регенерационных экосистем постиндустриальных ландшафтов был выбран один из золоотвалов ТЭЦ 5 г.Новосибирска (54°59' с.ш., 83°03' в.д.), а в качестве фонового (контрольного) участка, характеризующего данную территорию в природно-экологическом отношении, был выбран лесной биогеоценоз, расположенный недалеко от золоотвала (55°00' с.ш., 83°04' в.д.) в естественном березовом колке. Причиной, обусловившей данный выбор, являлось то, что на прилегающих территориях, ранее вовлеченных в сельскохозяйственный оборот, в настоящее время интенсивно протекает естественный процесс лесовосстановления. Таким образом, можно предположить, что конечной стадией направления регенерации при ряде условий может являться формирование на поверхности золоотвала именно лесных сообществ. Оба объекта расположены в лесостепи. Район их местонахождения теплый, с суммой среднесуточных температур выше 10 °С 1926 °С·день средняя температура июля составляет 25,7°C, и умеренно влажный, с годовым количеством осадков 300–400 мм; безморозный период длится 12-130 дней (Климат…, 2014).
Эксплуатация выбранного для исследования гидроотвала была прекращена в 2008 году, до 2010 года для снижения пыления на его поверхности постоянно поддерживали зеркало воды. Материнская порода представлена зольным материалом, на поверхности которого идет процесс самозарастания. Площадь гидроотвала составляет 20 га. Почвенный покров данного участка представлен эмбриоземом инициальным и эмбриоземом органо-аккумулятивным в соответствии с с классификацией Андроханова и др. (2004), или Technosols в соответствии с международной классификацией (IUSS Working Group WRB, 2014). Образцы эмбриозема органо-аккумулятивного, отбирали для данной работы. На поверхности сформировалась открытая растительная группировка – разнотравно-вейниковая с облепихой крушеновидной (Calamagrostis epigeios + Berteroa incana + Artemisia vulgaris – Hippophaë rhamnoides) с общим проективным покрытием составляло 3%, при средней высоте травостоя 40 см. Мхи покрывали 80% поверхности участка, в качестве доминантов были отмечены Leptobryum pyriforme (Hedw.) Wits., Bryum sp.; встречались также Funaria hydrometrica Hedw. и Bryum argenteum Hedw., а из древесных видов - подрост облепихи крушеновидной (Hippophaë rhamnoides L.) и тополя пирамидального (Populus italica Du Roi) (Sheremet et al., 2018).
Почва фонового участка классифицируется как серая (Классификация…, 2004), или как Gley-Luvic Phaeozem (IUSS Working Group WRB, 2015). Основной лесообразующей породой является береза белая (Betula pendula Roth.), но изредка попадаютря деревья сосны обыкновенной (Pinus sylvestris L.). Под покровом деревьев растет богатое злаково-разнотравное сообщество из 47 видов растений, относящихся к 27 семействам, в основном Fabaceae, Asteraceae,Caryophyllaceae, Plantaginaceae, Polygonaceae, Hypericaceae, Primulaceae, Lamiaceae, Euphorbiaceae, Rosaceae, Lythraceae, Apiaceae–Ubelliferae.
Отбор почвенных образцов. Почвенные образцы отбирали из 0-5 см слоя гумусово-аккумулятивного горизонта серой почвы под ненарушенным березовым лесом (контроль) и эмбриозема органо-аккумулятивного (золоотвал после 9 лет самозарастания) в конце июля 2017 г. в трех индивидуальных повторностях и сразу же доставляли в лабораторию. Почву просеивали через сито с диаметром ячеек 2 мм и до проведения физико-химических анализов хранили в холодильнике при +4 °С. Аликвоты почвы для экстракции ДНК хранили при -20 °С.
Химические анализы. Содержание Сорг определяли путем бихроматного окисления аликвоты почвенного образца. Определение содержания органического азота в почве проводили по Кьельдалю; содержание подвижных форм питательных элементов (NO3-, NH4+, P2O5), водорастворимого углерода (Свод) и рН (Н2О) измеряли стандартными методами (Soil Sampling…, 2008). Все анализы выполняли в трехкратной повторности. Базальное дыхание почвы (CO2) определяли путем измерения выделения СО2 почвой после стабилизации и без каких-либо добавок (Pell et al., 2006). Все значения рассчитаны на абсолютно-сухую почву и представлены в табл.1.
Таблица 1. Некоторые физико-химические свойства серой почвы и эмбриозема самозарастающего золоотвала
| Свойство | Серая почва | Эмбриозем | p* | ||
|---|---|---|---|---|---|
| Удельная масса, г·см-3 почвы | 0,42 | ± 0,02 | 1,31 | ± 0,06 | 0,00 |
| pH | 6,5 | ± 0,04 | 8,5 | ± 0,09 | 0,00 |
| Свод**, мкг·кг-1 почвы | 56 | ± 13 | 22 | ± 1 | 0,00 |
| Cорг ,% | 7,51 | ± 0,05 | 1,61 | ± 0,09 | 0,00 |
| Nорг, % | 0,43 | ± 0,01 | 0,04 | ± 0,00 | 0,00 |
| Cорг/Nорг | 20,3 | ± 0,3 | 65 | ± 1,4 | 0,00 |
| NO3-, мг N·кг-1 почвы | 2,9 | ± 0,1 | 1,9 | ± 0,1 | 0,00 |
| NH4+, мг N·кг-1 почвы | 1,7 | ± 0,6 | 1,9 | ± 0,1 | 0,20 |
| Подвижный P2О5, мг·кг-1 почвы | 1,2 | ± 0,0 | 6,1 | ± 0,1 | 0,00 |
| Pобщ, мг·кг-1 почвы | 84,9 | ± 0,6 | 47 | ± 2,7 | 0,00 |
| CO2, мкл · ч-1 · г-1 почвы | 11,2 | ± 0,9 | 0,5 | ± 0,1 | 0,00 |
* Вероятность отсутствия различий (t-критерий Стьюдента)
** Обозначения: Cорг – водорастворимый органический C, Cорг – почвенный органический C, Nорг – почвенный органический N
Экстракция ДНК из почвы. ДНК экстрагировали из почвы с помощью набора DNeasy PowerSoil Kit (Qiagen, Германия) в соответствии с инструкцией производителя. Для лучшего механического разрушения образца использовали TissueLyser II (Qiagen, Германия) в течение 45 с при 5000 об/мин. Дополнительной очистки извлеченной ДНК не требовалось; качество ее выделения оценивали с помощью электрофореза в 1%-ном агарозном геле и тестовой ПЦР.
Амплификация и секвенирование генов 16S рРНК. Экстрагированную из почв метагеномную ДНК использовали в качестве матрицы для амплификации вариабельного района (V3-V4) генов 16S рРНК с помощью праймеров 343F (5’-CTCCTACGGRRSGCAGCAG-3’) и 806R (5’-GGACTACNVGGGTWTCTAAT-3’) в сочетании с адаптерными последовательностями Illumina, а также баркодами на праймерах. Полимеразную цепную реакцию проводили в трех повторностях в объеме 50 мкл (Kirillova et al., 2016). Библиотеки секвенировали с помощью 600-cycle PE kit на секвенаторе MiSeq (Illumina, США) в ЦКП «Геномика» (ИХБФМ СО РАН, г.Новосибирск).
Биоинформатический анализ. Полученные парные последовательности анализировали с помощью UPARSE скриптов (Edgar, 2013), используя Usearch v.10.0.240 (Edgar, 2010). Биоинформатическая обработка включала перекрывание парных ридов, фильтрацию по качеству и длине, учет одинаковых последовательностей, отбрасывание синглетонов, удаление химер и объединение в операционные таксономические единицы (ОТЕ, в метагеномных исследованиях применяется в качестве синонима понятию вида). Последнее проводили на основе 97%-ного порога сходства ОТЕ с помощью алгоритма кластеризации UNOISE (Edgar, 2016а). Таксономическую принадлежность последовательностей ОТЕ определяли с помощью SINTAX (Edgar, 2016b) по базе рибосомальных последовательностей RDP.
Таксономическую структуру совокупности полученных таким образом последовательностей бактериального ансамбля оценивали по отношению числа последовательностей каждого таксона к общему числу последовательностей, выраженному в процентах, т.е. по относительному обилию таксона. Доминантными считали таксоны с относительным обилием ≥1%; умеренными и крупными доминантами считали таксоны с относительным обилием от 1 до 5% и свыше 5%, соответственно. Термин «ансамбль» (английский эквивалент assemblage), в соответствии с предложением Фота и др. (Fauth et al., 1996), использован как обозначающий филогенетически родственные группы в пределах сообщества, т.е. совокупности разных видов, встречающихся в одном месте в одно время.
Статистическую обработку данных проводили методами описательной статистики, корреляционного анализа и анализа главных компонент с помощью пакета Statistica v.13.3. Индексы биоразнообразия рассчитывали с помощью статистического пакета PAST 3.19 (Hammer et al., 2001).
Результаты исследования
Видовое богатство бактериальных ансамблей. Всего в данном исследовании было выявлено 4887 различных ОТЕ (Табл.2), из которых подавляющее большинство принадлежало 26 типам Bacteria, а 35 ОТЕ принадлежали представителям 6 типов Archaea.
Представители типа Proteobacteria доминировали по видовому богатству (1421 ОТЕ), составляя 29% от числа выявленных ОТЕ. У приблизительно такой же части всех ОТЕ, а именно 1171 ОТЕ, т.е. 24%, типовую принадлежность установить не удалось. К типу Acidobacteria было отнесено 425 ОТЕ, а к типу Actinobacteria – 443 ОТЕ, что в обоих случаях составляет 9% от общего богатства ОТЕ в нашем исследовании.
Таблица 2. Таксономический состав бактериальных ОТЕ в образцах контрольной серой почвы и самозарастающего золоотвала
| Таксон | Все ОТЕ | Доминантные ОТЕ |
|---|---|---|
| Надцарство | 2 | 1 |
| Тип | 32 | 7 |
| Класс | 84 | 13 |
| Порядок | 123 | 9 |
| Семейство | 212 | 19 |
| Род | 306 | 20 |
| ОТЕ | 4887 | 33 |
Относительное обилие таксонов в бактериальных ансамблях. Так же, как и по богатству ОТЕ, по относительному обилию последовательностей фрагментов генов 16S рРНК в почвах обеих экосистем доминировали представители Proteobacteria, составляя более трети всего ансамбля последовательностей (рис.1). По многим таксонам была выявлена статистически значимая разница между изученными экосистемами (табл.3-5). Относительное обилие представителей порядков Rhizobiales, Actinomycetales и неклассифицированных представителей Acidobacteria_Gp6 и Acidobacteria_Gp3 было в 1,5-2,4 раза больше в контрольной почве; при этом и на золоотвале они были основными доминирующими порядками (табл. 5).
Так же, как и по числу ОТЕ, по относительному обилию большая доля бактериальных последовательностей (рис.1), особенно в эмбриоземе, не была идентифицирована даже до типового уровня.
Анализ структуры взаимоотношений почвенных образцов и относительного обилия типов бактерий в них (рис.2) методом главных компонент выявил разделение изученных экосистем по главной компоненте 1 (отвечающей за более двух третей общей дисперсии): положение образцов серой почвы определяло относительное обилие Acidobacteria и Actinobacteria, а образцов эмбриозема – Chloroflexi и неклассифицированные Bacteria. Главная компонента 2, отвечающая за пятую часть общей дисперсии, выявила разброс между индивидуальными повторностями в пределах обеих групп, особенно выраженный в ненарушенной почве и связанный с обильностью Actinobacteria. Корреляционный анализ показал, что относительное обилие Acidobacteria статистически значимо связано с содержанием Nорг в почве, выделением СО2 (для обоих показателей Rспирмена=0,83, P≤ 0,05) и отношением C/N (Rспирмена=-0,83, P≤ 0,05), а относительное обилие Chloroflexi имеет противоположный этому корреляционный профиль. Среди умеренных доминантов Gemmatimonadetes имели положительную корреляцию с плотностью почвы (Rспирмена=0,94, P≤ 0,05), а обилие Verrucomicrobia статистически значимо не связано с почвенными свойствами, хотя и вносит вклад в дифференциацию образцов по обоим главным компонентам.
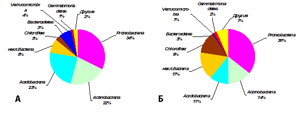
Рисунок 1. Таксономическая структура бактериальных ансамблей в образцах серой почвы под березовым лесом и эмбриозема самозарастающего золоотвала (оцененная по вкладу тип-специфичных последовательностей генов 16S рРНК в общее число последовательностей): А – лес, Б – золоотвал. Обозначения: некл.Bacteria - последовательности, относящиеся к царству Bacteria, но не идентифицированные даже до уровня типа. Числа представляют среднее значением (n=3).
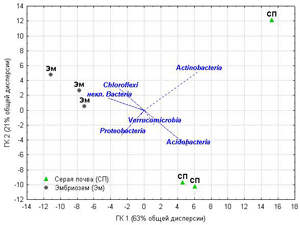
Рисунок 2.Анализ методом главных компонент относительного обилия тип-специфичных последовательностей в бактериальных ансамблях образцов серой почвы под березовым лесом и эмбриозема самозарастающего золоотвала. Обозначения: некл.Bacteria - последовательности, относящиеся к Bacteria, но не идентифицированные ниже уровня типа
Таблица 3. Относительное обилие тип-специфичных последовательностей фрагментов генов 16S рРНК в серой почве под березовым лесом (контроль) и эмбриоземе самозарастающего золоотвала ТЭЦ (среднее ± стандартное отклонение)
| Тип | Контроль | Золоотвал | p* |
|---|---|---|---|
| Proteobacteria | 32,6± 6,3 | 35,6± 3,0 | 0,50 |
| Actinobacteria | 21,6± 11,4 | 14,1± 1,6 | 0,32 |
| Bacteroidetes | 3,3± 1,2 | 3,5± 0,7 | 0,85 |
| Acidobacteria** | 23,2± 4,4 | 11,2± 0,6 | 0,01 |
| Verrucomicrobia | 4,0± 2,4 | 0,9± 0,2 | 0,09 |
| Candidatus WPS-1 | 0,3± 0,0 | 0,2± 0,1 | 0,05 |
| некл.Bacteria | 8,4± 0,1 | 16,6± 1,1 | 0,00 |
| Chloroflexi | 3,4± 2,3 | 9,5± 4,1 | 0,09 |
| Gemmatimonadetes | 1,3± 0,3 | 2,1± 0,4 | 0,04 |
| Candidatus Saccharibacteria | 0,6± 0,1 | 1,5± 0,1 | 0,01 |
| Nitrospirae | 0,2± 0,1 | 1,1± 0,1 | 0,00 |
| Firmicutes | 0,2± 0,1 | 0,6± 0,1 | 0,00 |
| Другие | 3,7± 0,8 | 9,0± 1,2 | 0,08 |
| Euryarchaeota | 0,00± 0,0 | 1,5± 0,1 | 0,00 |
* Вероятность отсутствия различий (t-критерий Стьюдента)
** жирным шрифтом выделены строки, где различия статистически значимы на уровне P≤0,05, а разным фоном выделены строки с отсутствием различий (желтый фон), с более высокими значениями в контрольной почве (зеленый фон) и с более высокими значениями на золоотвале (серый фон).
Таблица 4. Относительное обилие класс-специфичных последовательностей фрагментов генов 16S рРНК бактерий в серой почве под березовым лесом (контроль) и эмбриоземе самозарастающего золоотвала ТЭЦ (среднее ± стандартное отклонение)
| Класс | Контроль | Золоотвал | p* |
|---|---|---|---|
| Actinobacteria | 19,8± 10,6 | 13,9± 1,6 | 0,39 |
| unc_Chloroflexi | 1,6± 1,2 | 3,8± 2,0 | 0,18 |
| Cytophagia | 1,0± 0,3 | 1,2± 0,3 | 0,39 |
| Acidobacteria_Gp17 | 0,9± 0,4 | 1,2± 0,2 | 0,25 |
| Sphingobacteriia | 1,2± 0,6 | 0,9± 0,3 | 0,55 |
| Alphaproteobacteria | 18,8± 4,0 | 11,3± 0,6 | 0,03 |
| Acidobacteria_Gp6 | 13,3± 3,7 | 5,9± 0,4 | 0,03 |
| Acidobacteria_Gp16 | 2,7± 0,6 | 1,6± 0,3 | 0,05 |
| Spartobacteria | 3,4± 2,3 | 0,1± 0,0 | 0,06 |
| Gemmatimonadetes | 1,3± 0,3 | 2,1± 0,4 | 0,05 |
| Acidobacteria_Gp3 | 2,0± 0,1 | 0,9± 0,1 | 0,00 |
| Acidobacteria_Gp4 | 1,9± 0,5 | 0,9± 0,1 | 0,03 |
| некл.Bacteria | 8,4± 0,1 | 16,6± 1,1 | 0,00 |
| Betaproteobacteria | 5,3± 1,1 | 10,8± 1,9 | 0,01 |
| Deltaproteobacteria | 5,2± 0,3 | 6,5± 0,6 | 0,02 |
| Gammaproteobacteria | 3,0± 1,0 | 6,4± 0,1 | 0,00 |
| Anaerolineae | 1,4± 1,0 | 5,2± 2,1 | 0,04 |
| Другие | 8,9± 0,2 | 10,2± 0,1 | 0,00 |
* Вероятность отсутствия различий (t-критерий Стьюдента)
** жирным шрифтом выделены строки, где различия статистически значимы на уровне P≤0,05, а разным фоном выделены строки с отсутствием различий (желтый фон), с более высокими значениями в контрольной почве (зеленый фон) и с более высокими значениями на золоотвале (серый фон).
Таблица 5. Относительное обилие порядок-специфичных последовательностей фрагментов генов 16S рРНК бактерий в серой почве под березовым лесом (контроль) и эмбриоземе самозарастающего золоотвала ТЭЦ (среднее ± стандартное отклонение)
| Порядок | Контроль | Золоотвал | p* |
|---|---|---|---|
| некл.Betaproteobacteria | 3,8± 0,8 | 5,5± 1,1 | 0,09 |
| некл.Actinobacteria | 4,9± 1,9 | 3,4± 0,5 | 0,28 |
| Myxococcales | 4,1± 0,3 | 3,4± 0,5 | 0,14 |
| Rhodospirillales | 3,6± 1,2 | 2,2± 0,2 | 0,12 |
| некл.Chloroflexi | 1,6± 1,1 | 3,8± 2,0 | 0,18 |
| Cytophagales | 1,0± 0,3 | 1,2± 0,3 | 0,39 |
| некл.Acidobacteria_Gp17 | 0,9± 0,4 | 1,2± 0,2 | 0,25 |
| Sphingobacteriales | 1,2± 0,6 | 0,9± 0,3 | 0,55 |
| некл.Acidobacteria_Gp6 | 13,3± 3,7 | 5,9± 0,4 | 0,03 |
| Rhizobiales | 12,6± 1,8 | 5,8± 0,3 | 0,00 |
| Actinomycetales | 10,6± 8,2 | 4,2± 0,4 | 0,25 |
| некл.Acidobacteria_Gp16 | 2,6± 0,6 | 1,6± 0,3 | 0,05 |
| некл.Spartobacteria | 3,4± 2,3 | 0,1± 0,0 | 0,06 |
| некл.Acidobacteria_Gp3 | 2,0± 0,1 | 1,3± 0,2 | 0,00 |
| некл.Acidobacteria_Gp4 | 1,8± 0,5 | 0,9± 0,1 | 0,03 |
| Gaiellales | 2,1± 0,2 | 0,4± 0,2 | 0,00 |
| некл.Bacteria | 8,4± 0,1 | 16,6± 1,1 | 0,00 |
| Acidimicrobiales | 3,6± 1,3 | 5,9± 0,7 | 0,06 |
| Anaerolineales | 1,4± 1,0 | 5,2± 2,1 | 0,04 |
| Burkholderiales | 1,4± 0,4 | 4,3± 0,8 | 0,01 |
| Xanthomonadales | 1,5± 0,4 | 3,4± 0,2 | 0,00 |
| Gemmatimonadales | 1,3± 0,3 | 2,1± 0,4 | 0,04 |
| некл.Alphaproteobacteria | 0,9± 0,3 | 2,1± 0,2 | 0,00 |
| некл.Gammaproteobacteria | 1,2± 0,7 | 1,5± 0,3 | 0,06 |
| Desulfuromonadales | 0,1± 0,1 | 2,6± 1,9 | 0,02 |
| некл.Candidatus Saccharibacteria | 0,6± 0,1 | 1,3± 0,6 | 0,01 |
| Другие | 11,2± 3,0 | 13,2± 2,5 | 0,04 |
* Вероятность отсутствия различий (t-критерий Стьюдента)
** жирным шрифтом выделены строки, где различия статистически значимы на уровне P≤0,05, а разным фоном выделены строки с отсутствием различий (желтый фон), с более высокими значениями в контрольной почве (зеленый фон) и с более высокими значениями на золоотвале (серый фон).
Таблица 6. Относительное обилие доминантных OTЕ-специфичных последовательностей фрагментов генов 16S рРНК бактерий серой почве под березовым лесом (контроль) и эмбриоземе самозарастающего золоотвала ТЭЦ (среднее ± стандартное отклонение)
| № | OTU | Контроль | Золоотвал | p* |
|---|---|---|---|---|
| 9 | Mycobacterium sp | 2,5± 1,9 | 0,1± 0,0 | 0,10 |
| 54 | некл.Chloroflexi | 0,4± 0,6 | 0,7± 0,6 | 0,52 |
| 3 | Bradyrhizobium sp.** | 6,4± 1,1 | 0,2± 0,1 | 0,00 |
| 16 | некл.Acidobacteria_Gp16 | 1,2± 0,3 | 0,0± 0,0 | 0,01 |
| 20 | некл.Rhizobiales | 2,3± 0,3 | 0,3± 0,1 | 0,00 |
| 22 | Spartobacteriagis*** | 1,6± 1,0 | 0,0± 0,0 | 0,05 |
| 24 | Ilumatobacter sp. | 0,7± 0,2 | 0,3± 0,1 | 0,04 |
| 33 | Reyranella sp. | 0,8± 0,3 | 0,3± 0,0 | 0,03 |
| 51 | некл.AcidobacteriaGp6 | 0,8± 0,2 | 0,4± 0,1 | 0,03 |
| 63 | некл.AcidobacteriaGp6 | 1,0± 0,3 | 0,0± 0,0 | 0,01 |
| 71 | некл.AcidobacteriaGp6 | 0,9± 0,3 | 0,0± 0,0 | 0,01 |
| 121 | некл.AcidobacteriaGp6 | 1,3± 0,5 | 0,0± 0,0 | 0,01 |
| 27 | некл.Anaerolineaceae | 0,1± 0,1 | 2,4± 1,6 | 0,07 |
| 28 | некл.Chloroflexi | 0,1± 0,1 | 2,5± 1,3 | 0,03 |
| 30 | некл.Xanthomonadales | 0,2± 0,1 | 1,3± 0,2 | 0,00 |
| 31 | Geobacter sp. | 0,0± 0,0 | 1,0± 0,6 | 0,05 |
| 34 | некл.Actinobacteria | 0,0± 0,0 | 1,1± 0,1 | 0,00 |
| 36 | некл.AcidobacteriaGp6 | 0,0± 0,0 | 1,4± 0,1 | 0,00 |
| 42 | некл.Actinobacteria | 0,0± 0,0 | 0,7± 0,2 | 0,02 |
| 45 | Nitrospira sp. | 0,0± 0,0 | 0,9± 0,1 | 0,00 |
| 46 | некл.AcidobacteriaGp6 | 0,3± 0,0 | 1,0± 0,1 | 0,00 |
| 56 | Aciditerrimonas sp. | 0,0± 0,0 | 1,1± 0,2 | 0,00 |
| 61 | некл.Iamiaceae | 0,4± 0,2 | 1,0± 0,2 | 0,04 |
| 119 | некл.Bacteria | 0,0± 0,0 | 0,7± 0,3 | 0,02 |
| 134 | Rhodoferax sp. | 0,0± 0,0 | 1,0± 0,5 | 0,03 |
| 428 | некл.AcidobacteriaGp 16 | 0,2± 0,1 | 1,1± 0,2 | 0,01 |
| Другие | 77,8± 0,7 | 81,5± 1,8 | 0,03 |
* Вероятность отсутствия различий (t-критерий Стьюдента)
** жирным шрифтом выделены строки, где различия статистически значимы на уровне P≤0,05, а разным фоном выделены строки с отсутствием различий (желтый фон), с более высокими значениями в контрольной почве (зеленый фон) и с более высокими значениями на золоотвале (серый фон).
Индексы α-биоразнообразия ансамблей последовательностей генов 16SрРНК
Индексы α-биоразнообразия, характеризующие видовое богатство в каждой экосистеме (Као-1, число ОТЕ, Маргалефа, Фишера) были выше в контрольной почве (табл.7). Также выше были и индексы, отражающие большее доминирование (доминирование, индекс Бергера-Паркера). Индексы выравненности и равномерности распределения последовательностей по различным ОТЕ, наоборот, были выше на золоотвале, что, как правило, интерпретируют как большее биоразнообразие. Примечательно, что индекс Шеннона, который чаще всего применяют для оценки α-биоразнообразия, будучи очень высоким в почве обеих экосистем, достоверно не различался, так же как и индексы Симпсона, Бриллуэна и Менхиника. Таким образом, видовое (т.е. ОТЕ) богатство в почве золоотвала было на треть ниже по сравнению с таковым в контрольной почве, однако в пределах этого меньшего числа видов структура сообщества была более разнообразна.
Таблица 7. Индексы α-биоразнообразия (среднее ± стандартное отклонение) бактериальных ансамблей в гумусово-аккумулятивном горизонте лесной почвы под березовым лесом (контроль) и верхнем слое почвосубстрата самозарастающего золоотвала ТЭЦ (среднее ± стандартное отклонение)
| Индекс | Контроль | Золоотвал | p* |
|---|---|---|---|
| Симпсона | 0,993± 0,000 | 0,995± 0,001 | 0,07 |
| Шеннона | 6,3± 0,1 | 6,3± 0,1 | 0,64 |
| Бриллуэна | 6,2± 0,0 | 6,2± 0,1 | 0,88 |
| Менхиника | 9,8± 1,9 | 10,0± 0,4 | 0,84 |
| Число ОТЕ | 2910± 278 | 2166± 69 | 0,01 |
| Доминирование | 0,007± 0,000 | 0,005± 0,001 | 0,07 |
| Маргалефа | 254± 12 | 201± 5 | 0,00 |
| Фишера | 576± 13 | 470± 12 | 0,00 |
| Бергера-Паркера | 0,052± 0,006 | 0,026± 0,013 | 0,04 |
| Као-1 | 3356± 144 | 2391± 57 | 0,00 |
| Выравненность | 0,18± 0,03 | 0,25± 0,01 | 0,02 |
| Равномерность | 0,79± 0,02 | 0,82± 0,01 | 0,03 |
* Вероятность отсутствия различий (t-критерий Стьюдента).
** Жирным шрифтом выделены строки, где различия статистически значимы на уровне P≤0,05, а разным фоном выделены строки с отсутствием различий (желтый фон), с более высокими значениями в контрольной почве (зеленый фон) и с более высокими значениями на золоотвале (серый фон)
Обсуждение
Изучению развития экосистем отработанных золоотвалов посвящено много работ (Haynes, 2009), однако исследований, выполненных с помощью метагеномных методов, до сих пор очень мало. В большинстве работ разнообразие почвенных микроорганизмов, чаще всего бактерий, оценено с помощью методов культивирования на твердых питательных средах (см., например, Pen-Muratov et al., 2014); хотя давно понятно, что эти методы неадекватны целям оценки состава и структуры сообществ микроорганизмов (McCaig et al., 1999).
За девять лет самозарастания золоотвала в формирующемся эмбриоземе развился богатый видами выровненный по структуре бактериальный ансамбль, который на этом раннем этапе педогенеза на уровне типа был уже близок к таковому в серой почве под ненарушенным лесом, расположенном по соседству с золоотвалом. На уровне типа основные доминанты бактериальных ансамблей эмбриозема и серой почвы были одинаковы (Proteobacteria, Acidobacteria и Actinobacteria), и по общему очертанию относительного обилия типов изученные бактериальные ансамбли были довольно схожи, что в целом свидетельствует о возможности направленности почвообразования к серой почве. Основное отличие касалось Acidobacteria – главного доминанта почв разнообразных лесных экосистем мира наряду с Proteobacteria. Представители последнего доминировали в изученных почвах как по видовому разнообразию, так и по относительному обилию, которое было одинаково в обеих почвах, что в общем согласуется с результатами других исследований (Chen et al., 2017б). В этой же работе выявлено более низкое биоразнообразие бактериального ансамбля зарастающего золоотвала, при этом величины индекса α–биоразнообразия Шеннона как в контроле, так и на золоотвале были в 2,5−3,0 раза ниже рассчитанных для наших объектов (6,2-6,3). Примечательно, что в нашей работе выравненность и равномерность бактериального ансамбля эмбриозема были больше, чем в серой почве, что может свидетельствовать о том, что большее число организмов опробует разнообразие микрообитаний на начальных стадиях педогенеза.
Тем не менее, на более низких таксономических уровнях бактериальный ансамбль эмбриозема существенно отличался от такового в ненарушенной серой почве. Значительно большее относительное обилие в эмбриоземе представителей типа Chloroflexi согласуется с имеющимися сведениями об их физиологии и биохимии, т.е. что среди них есть аэробные и анаэробные термофилы, аноксигенные фототрофы и организмы, использующие органогалиды в качестве субстратов для анаэробного дыхания (Krzmarzick et al., 2012), а также о том, что обилие представителей типа Chloroflexi увеличивается при орошении сточными водами (Chen et al., 2017а). Таким образом, значительно повышенное присутствие представителей типа Chloroflexi в молодом эмбриоземе самозарастающего в течение девяти лет зольного гидроотвала вполне логично с точки зрения экологических условий формирования отвала (искусственный водоем) и его последующего самозарастания (открытая поверхность с резкими колебаниями температуры). Повышенное обилие одного вида, представляющего семейство Anaerolineaceae (тип Chloroflexi), в эмбриоземе обусловлено, скорее всего, щелочным pH (Guo et al., 2019).
Среди бактерий, относящихся к порядку Xanthomonadales (класс Gammaproteobacteria), наряду с патогенами растений и человека, есть много свободноживущих непатогенных видов, способных активно разлагать целлюлозу и выживать в таких неблагоприятных местообитаниях, как загрязненные почвы и горячие источники (Eichorst, Kuske, 2012; Castro-Silva et al., 2013; Bayer-Santos et al., 2019), и поэтому их присутствие среди доминантов эмбриозема также выглядит вполне обоснованным экофизиологически.
Род Geobacter объединяет очень интересные бактерии с уникальной физиологией (Lovley et al., 2011; Lovley, Walker, 2019), способные, в частности, восстанавливать нерастворимые оксиды Fe (III) и Mn (IV), т.е. осуществлять процессы огромного биогеохимического значения, и присутствие Geobacter sp. среди доминантов в эмбриоземе, развивающемся на зольном субстрате в качестве педогенной матрицы, говорит само за себя. Восстанавливать Fe (III), а также детоксицировать и/или использовать в качестве углеродных субстратов органические соединения антропогенного происхождения (например, при неполном сжигании углей и/или загрязнении речной воды, используемой для гидротранспортировки), могут и бактерии рода Rhodoferax (Kasanke et al., 2019), одна ОТЕ которого была выявлена среди доминантов бактериального ансамбля эмбриозема в нашем исследовании. Что касается Aciditerrimonas (тип Actinobacteria), то представители этого рода тоже способны восстанавливать Fe (III) в анаэробных условиях (Itoh et al., 2011). Большее обилие одной относящейся к семейству Iamiaceae ОТЕ (тип Actinobacteria) в эмбриоземе согласуется со способностью этих бактерий жить в условиях повышенного содержания металлов, активно участвуя в становлении и поддержании процессов трансформации абиотической и биотической составляющих педогенного субстрата. Таким образом, на ранних стадиях преобразования зольного субстрата в бактериальном ансамбле молодого эмбриозема доминирует специфичный комплекс бактерий, осуществляющих выветривание материнской породы.
Несмотря на то, что четыре ОТЕ, отнесенные к классу Acidobacteria Gp6, были более обильны в ненарушенной почве, две других ОТЕ того же класса были более обильны в эмбриоземе. В целом это свидетельствует о разнообразии экологических ниш бактерий этого класса и о важности их роли в почвах наземных экосистем в целом и в ходе педогенеза в техногенных экосистемах в частности.
Доминирование представителей порядка Rhizobiales, очень существенное в изученной нами серой почве и в целом типичное для почв лесных экосистем (Naumova et al., 2015; Наумова и др., 2018; VanInsberghe et al., 2015; Dukunde et al., 2019), было выявлено и в эмбриоземе, хотя и значительно менее выраженное. Это свидетельствует об исключительной важности процесса фиксации атмосферного азота для становления цикла азота и формирования биогеоценоза на золоотвале.
Самым крупным доминантом в серой ненарушенной почве была одна ОТЕ, представляющая род Bradyrhizobium. Ранее установлено, что несимбиотические, т.е. свободно живущие представители этого рода доминируют в лесных почвах (VanInsberghe et al., 2015). Следовательно, выделение этой бактерии в культуре и изучение ее физиолого-биохимических особенностей очень целесообразно для разработки препарата этой бактерии для внесения в отработанные золоотвальные субстраты и ускорения восстановления экосистемных функций и сервисов при рекультивационных технологиях.
Присутствие среди доминантов эмбриозема одного из видов Nitrospira – широко распространенных в различных экосистемах бактерий (Daims et al., 2016) – говорит о формировании связи между восстановленными и окисленными формами минерального азота (Palomo et al., 2019). Одна ОТЕ рода Reyranella, представители которого являются денитрификаторами и обнаружены в лесных почвах (Kim et al., 2013), была выявлена и в эмбриоземе. Все это вместе с доминированием порядка Rhizobiales свидетельствует о становлении всего комплекса процессов трансформации азота уже на ранних стадиях педогенеза на самозарастающем золоотвале.
Следует заметить, что повышенное обилие Nitrospira, Geobacter, Actinobacteria и Acidobacteria в эмбриоземе может указывать на наличие тяжелых металлов, в частности, меди (Nunes et al., 2016) в их окружении.
Представители Spartobacteria были среди основных доминантов с большим обилием в серой почве. Эти бактерии являются важными компонентами бактериальных ансамблей различных почв (Bergmann et al., 2011), осуществляющими разложение растительного материала (Ranjan et> al., 2015). Информации о физиологии и биохимии Spartobacteria до сих пор немного; было выдвинуто предположение об их меньшем метаболическом разнообразии и большей специализации (низкомолекулярные) в плане субстратов углерода и энергии для обеспечения доминирования в почве (Brewer et al., 2017). Таким образом, значительно меньшая представленность этого класса в эмбриоземе самовосстанавливающейся экосистемы золоотвала вероятнее всего связана с недостаточным поступлением нужного растительного материала и/или продуктов его гидролиза. Род Ilumatobacter, также более обильный в фоновой почве, плохо переносит присутствие металлов в среде обитания (Berg et al., 2019). Род Reyranella, среди которых есть денитрификаторы, встречается в лесных почвах (Kim et al., 2013), и один его вид является умеренным доминантом и в изученной нами серой почве; выявление его и на золоотвале свидетельствует о наличии агентов процесса денитрификации в этой экосистеме, подтверждая, как отмечено выше, становление всего комплекса процессов трансформации азота.
Отметим, что потенциальное видовое богатство (т.е. Као-1) составило в ненарушенной серой почве 3356 ОТЕ и в эмбриоземе 2391 ОТЕ при экстракции тотальной ДНК из 0,1 г сырой почвы. При этом индекс биоразнообразия Шеннона был довольно высок и одинаков в почве обеих экосистем, что свидетельствует о быстром формировании разнообразного состава бактериальной составляющей педогенных и экосистемных процессов.
Наличие 8-10% последовательностей фрагментов генов 16S рРНК, неидентифицированных даже до таксономического уровня типа, является обычным для большинства верхних горизонтов/слоев почв, что связано как с принципиальной невозможностью охватить все разнообразие прокариот на планете, так и с методическими аспектами анализа, техническими аспектами пополнения баз данных и т.п. Однако 17% неидентифицированных последовательностей эмбриозема представляют довольно значительную величину, которая объясняется, по-видимому, меньшей изученностью такого рода почвосубстратов и, как следствие, отсутствием информации о генах 16S рРНК в базах данных, в данном случае RDP.
Заключение
Доминирование в формирующемся эмбриоземе бактерий, информация о последовательностях генов 16S рРНК которых отсутствует в соответствующих базах данных, подчеркивает необходимость более детального исследования почвенных микробных ансамблей, развивающихся в ходе педогенеза при самозарастании или рекультивации отвалов в связи с увеличением площадей, занятых такого рода техногенными объектами. Выявление доминирования азотфиксаторов как в ненарушенной лесной почве, так и в формирующемся эмбриоземе обуславливает необходимость детального изучения ансамблей симбиотических и несимбиотических азотфиксаторов и возможности их использования для ускорения рекультивации и фиторемедиации с целью восстановления экосистемных сервисов. Особого внимания в этой связи заслуживают бактерии, осуществляющие выветривание техногенных субстратов в ходе педогенеза.
Финансовая поддержка
Работа по отбору и химическому анализу почвенных образцов, а также написанию статьи выполнена при поддержке государственного задания ИПА СО РАН в рамках программы с № госрегистрации АААА-А17-117030110078-1. Работа по секвенированию ДНК и биоинформатическому анализу выполнена при финансовой поддержке РФФИ и Правительства Новосибирской области в рамках Проекта №18-44-540002
Литература
- Андроханов В.А., Куляпина Е.Д., Курачев В.М. Почвы техногенных ландшафтов: генезис и эволюция. Новосибирск: Изд-во СО РАН, 2004. 205 с.
- Калачёв А. И. Комплексная система утилизации ЗШМ // Консорциум Феникс, июнь 2017. С. 3
- Классификация и диагностика почв России / Составители: Шишов Л.Л.,Тонконогов В.Д., Лебедева И.И., Герасимова М.И. Смоленск: Ойкумена, 2004. 342 с.
- Климат Новосибирска и его изменения / Лучицкая И.О., Белая Н.И., Арбузов С.А. Федеральная служба по гидрометеорологии и мониторингу окружающей среды, Сибирский региональный научно-исследовательский гидрометеорологический институт. Новосибирск: Издательство СО РАН, 201 224 с.
- Наумова Н. Б., Аликина Т. Ю., Кузнецова Г. В. Биоразнообразие бактериальных ансамблей в бурозёме элювиированном под сосной корейской // Почвы и окружающая среда. 2018. Т.1. №3. С.151-169. CrossRef
- Шилов С.О., Шилова Е.А. Выбор возможного варианта расширения золоотвала на основании сметного расчета // Синергия наук. 2017. № 12. С. 821-827. Полный текст
- Энергетика и промышленность России. 2019. №5. (361) март 2019 года. Полный текст
- Bayer-Santos E., Ceseti L.de M., Farah C.S., Alvarez-Martinez C.E. Distribution, Function and Regulation of Type 6 Secretion Systems of Xanthomonadales // Frontiers in Microbiology. 2019. CrossRef
- Berg J., Brandt K.K., Al-Soud W.A., Holm P.E., Hansen L.H., Sørensen S.J., Nybroe O. Selection for Cu-tolerant bacterial communities with altered composition, but unaltered richness, via long-term Cu exposure // Applied and Environmental Microbiology. 2012. V.78(20). P. 7438–7446. CrossRef
- Bergmann G. T., Bates S.T., Eilers K.G., Lauber C.L., Caporaso J.G., Walters W.A., Knight R., Fierer N. The under-recognized dominance of Verrucomicrobia in soil bacterial communities // Soil Biology and Biochemistry. 2011. V.43. Is.7. P. 1450–1455. CrossRef
- Brewer T., Handley K., Carini P. et al. Genome reduction in an abundant and ubiquitous soil bacterium ‘Candidatus Udaeobacter copiosus’ // Nature Microbiology. 2017. V.2. art. 16198. CrossRef
- Castro-Silva C., Ruíz-Valdiviezo, V. M, Valenzuela-Encinas C.et al. The bacterial community structure in an alkaline saline soil spiked with anthracene // Electronic Journal of Biotechnology. 2013. V.16. No.5. CrossRef
- Chen L, Feng Q, Li C. et al. Impacts of aquaculture wastewater irrigation on soil microbial functional diversity and community structure in arid regions // Sci Rep. 2017а. V.7. №1. Art.11193. CrossRef
- Chen X.W., Wong J.T., Leung A.O., Ng C.W., Wong M.H. Comparison of plant and bacterial communities between a subtropical landfill topsoil 15years after restoration and a natural area // Waste Management. 2017b. V.63, №49-57. CrossRef
- Daims H., Lücker S., Wagner M. A New Perspective on Microbes Formerly Known as Nitrite-Oxidizing Bacteria // Trends in Microbiology. V.24. Is.9. P.699-712. CrossRef
- Dukunde A., Schneider D., Schmidt M.et al. Tree Species Shape Soil Bacterial Community Structure and Function in Temperate Deciduous Forests // Frontiers in microbiology. 2019. V.10. Art. 1519. CrossRef
- Edgar R.C. Search and clustering orders of magnitude faster than BLAST // Bioinformatics. 2010.V.26. Iss.19. P. 2460-2461. CrossRef
- Edgar R.C. UPARSE: highly accurate OTU sequences from microbial amplicon reads // Nature Methods. 2013. V.10. P. 996–998. CrossRef
- Edgar R.C. UNOISE2: Improved error-correction for Illumina 16S and ITS amplicon reads // bioRxiv. 2016a. CrossRef
- Edgar R. C. SINTAX, a Simple Non-Bayesian Taxonomy Classifier for 16S and ITS Sequences // bioRxiv. 2016b. CrossRef
- Eichorst S.A., Kuske C.R. Identification of Cellulose-Responsive Bacterial and Fungal Communities in Geographically and Edaphically Different Soils by Using Stable Isotope Probing // Applien and Environmental Microbiology. 2012. V.78. №7. P. 2316–2327. CrossRef
- Fauth E., Bernardo J., Camara M., Resetarits Jr. W.J., Van Buskirk J., McCollum S.A. Simplifying the Jargon of Community Ecology: A Conceptual Approach // The American Naturalist. 1996. V.147. P.282-286.
- Guo A., Ding L., Tang Z., Zhao Z., Duan G. Microbial response to CaCO3 application in an acid soil in southern China // Journal of Environmental Science (China). 2019. V.79. P.321-329. CrossRef
- Hammer O., Harper D.A.T., Ryan P.D. PAST: Paleontological Statistics Software Package for Education and Data Analysis // Palaeontologia Electronica. 2001. V.4. Iss.1. P.9.
- Haynes R.J. Reclamation and revegetation of fly ash disposal sites - Challenges and research needs // J Environ Manage. 2009. V.90. №1. P. 43-53. CrossRef
- Itoh T., Yamanoi K., Kudo T., Ohkuma M., Takashina T. Aciditerrimonas ferrireducens gen. nov., sp. nov., an iron-reducing thermoacidophilic actinobacterium isolated from a solfataric field // International Journal of Systematic and Evolutionary Microbiology. 2011. V.61(Pt 6). P.1281-1285. CrossRef
- IUSS Working Group, 2014. WRB, World Reference Base for Soil Resources 2015. International soil classification system for naming soils and creating legends for soil maps. FAO, Rome. 203 p.
- Kasanke C.P., Collins R.E., Leigh M.B. Identification and Characterization of a Dominant Sulfolane-Degrading Rhodoferax sp. via Stable Isotope Probing Combined with Metagenomics // Scientific Reports. 2019. V.9. 3121. CrossRef
- Kim S.J., Ahn J.H., Lee T.H., Weon H.Y., Hong S.B., Seok S.J., Whang K.S., Kwon S.W. Reyranella soli sp. nov., isolated from forest soil, and emended description of the genus Reyranella Pagnier et al. 2011 // International Journal of Systematic and Evolutionary Microbiology. 2013. V.63(Pt 9). P.3164-3167. CrossRef
- Kirillova I.V., Argant J., Lapteva E.G., Korona O.M., J. van der Plicht., Zinovyev E.V., Kotov A.A., Chernova O.F., Fadeeva E.O., Baturina O.A., Kabilov M.R., Shidlovskiy F.K., Zanina O.G. The diet and environment of mammoths in North-East Russia reconstructed from the contents of their feces // Quaternary International. 2016. V. 406. P. 147-161. CrossRef
- Krzmarzick M.J., Crary B.B., Harding J.J., Oyerinde O.O., Leri A.C., Myneni S.C., Novak P.J. Natural niche for organohalide-respiring Chloroflexi // Applied and Environmental Microbiology. 2012. V.78. №2. P. 393-401. CrossRef
- Lovley D.R., Walker D.J. F. Geobacter Protein Nanowires // Frontiers in Microbiology. 2019. V.10. P. 2078. CrossRef
- Lovley D.R., Ueki T., Zhang T., Malvankar N.S., Shrestha P.M., Flanagan K.A., Aklujkar M., Butler J.E., Giloteaux L., Rotaru A.E., Holmes D.E., Franks A.E., Orellana R., Risso C., Nevin K.P.. Geobacter: the microbe electric's physiology, ecology, and practical applications // Advances in Microbial Physiology. 2011. V.59. P.1-100. CrossRef
- McCaig A.E., Glover L.A., Prosser J.I. Molecular analysis of bacterial community structure and diversity in unimproved and improved upland grassland pastures // Applied and Environmental Microbiology. 1999. V. 65. №4. pp. 1721-1730.
- Naumova N.B., Kuznetsova G.V., Alikina T.Y., Kabilov M.R. Bacterial 16S DNA diversity in the rhizosphere soil of two pine species // Биомика. 2015. Т.7. № 2. С.127-136. Полный текст
- Nunes I., Jacquiod S., Brejnrod A., Holm P.E., Johansen A., Brandt K.K., Priemé A., Sørensen S.J. Coping with copper: legacy effect of copper on potential activity of soil bacteria following a century of exposure // FEMS Microbiology Ecology. 2016. V.92. №11. fiw175. CrossRef
- Palomo A., Dechesne A., Smets B.F. Genomic profiling of Nitrospira species reveals ecological success of comammox Nitrospira // bioRxiv. 2019. 612226. CrossRef
- Pell M., Stenstrom L., Granhall U. Soil respiration // Microbiological methods for Assessing Soil Quality. Wallingford: CABI International, 2005. P.117-126.
- Pen-Mouratov S., Shukurov N., Yu J., Rakhmonkulova S., Kodirov O., Barness G., Kersten M., Steinberger Y. Successive development of soil ecosystems at abandoned coal-ash landfills // Ecotoxicology. 2014. V.23(5). P.880-897. CrossRef
- Ranjan K., Paula F.S., Mueller R.C., Jesus Eda C., Cenciani K., Bohannan B.J., Nüsslein K., Rodrigues J.L. Forest-to-pasture conversion increases the diversity of the phylum Verrucomicrobia in Amazon rainforest soils // Frontiers in Microbiology. 2015. V.6. Art.779. CrossRef
- Sheremet N., Belanov I., Doronkin V., Lamanova T., Naumova N. Biogeocenosis development during initial revegetation of a coal combustion ash dump // BIO Web of Conferences 2018. 11. 00038 Prospects of Development and Challenges of Modern Botany. CrossRef
- Soil Sampling and methods of Analysis. 2nd edition. Boca Raton: CRC Press; 2008. 1224 p.
- VanInsberghe D., Maas K. R., Cardenas E., Strachan C. R., Hallam S. J., Mohn W.W. Non-symbiotic Bradyrhizobium ecotypes dominate North American forest soils // The ISME journal. 2015.V.9. №11. P. 2435-2441. CrossRef
- Yamada T., Sekiguchi Y. Cultivation of Uncultured Chloroflexi Subphyla: Significance and Ecophysiology of Formerly Uncultured Chloroflexi 'Subphylum I' with Natural and Biotechnological Relevance // Microbes and environments. JSME. 2009. V.24. P. 205-216. CrossRef
Сведения об авторах:
Наумова Наталья Борисовна – кандидат биологических наук, ведущий научный сотрудник лаборатории агрохимии Института почвоведения и агрохимии СО РАН (Новосибирск, Россия); naumova@issa-siberia.ru
Беланов Иван Петрович – кандидат биологических наук, старший научный сотрудник лаборатории рекультивации Института почвоведения и агрохимии СО РАН (Новосибирск, Россия); belanov@issa-siberia.ru
Аликина Татьяна Юрьевна – младший научный сотрудник ЦКП «Геномика» Института химической биологии и фундаментальной медицины СО РАН (Новосибирск, Россия); alikina@niboch.nsc.ru
Авторы прочитали и одобрили окончательный вариант рукописи
 Статья доступна по лицензии Creative Commons Attribution 4.0 License
Статья доступна по лицензии Creative Commons Attribution 4.0 License
TAXONOMIC DIVERSITY OF BACTERIAL ASSEMBLAGE IN TECHNOSOL OF THE REVEGETATING FLY ASH DUMP
N.B. Naumova1
 , I.P. Belanov1
, I.P. Belanov1
 , T.Y. Alikina2
, T.Y. Alikina2
Address: 1 Institute of Soil Science and Agrochemistry, Siberian Branch of the Russian Academy of Sciences, Novosibirsk, Russia
2Institute of Chemical Biology and Fundamental Medicine, Siberian Branch, Russian Academy of Sciences, Novosibirsk, Russia
Background. The aim of the study was to assess the composition and structure of bacterial assemblages by estimating 16S rRNA gene sequences diversity in a young Technosol, developing on a revegetating fly ash dump.
Location and place of the study. Two soils were chosen for the study in the environs of the thermoelectric power station No.5 in Novosibirsk region: 1) nine years old Technosol (54°59' NL, 83°03' EL), developing on the spontaneously revegetating fly ash dump, and 2) Phaeozem under undisturbed white birch forest (55°00' NL, 83°04' EL). Soils were sampled from the 0-5 cm layer as three individual replicates each.
Materials and methods. The total DNA extracted from soil samples was used as a matrix for PCR amplification using V-3-V4 primers for 16S rRNA genes. The obtained amplicons were sequenced on Illumina MiSeq platform in the Genomics Core Facility of the Institute of Chemical Biology and Fundamental Medicine SB RAS (Novosibirsk, Russia). Descriptive statistics and principal components analysis were used to analyze and present the data.
Results. Overall 4887 different operational taxonomic units (OTUs) were found at the 97% similarity level; the OTUs were ascribed to 306 genera, 212 families, 123 orders, 84 classes and 32 phyla of bacteria domain. At the phylum level bacterial assemblages in both soils were dominated by Proteobacteria, Acidobacteria and Actinobacteria, showing rather similar outline of the phyla relative abundance, confirming the direction of pedogenesis towards Phaeozem. Higher abundance of Chloroflexi a physiologically diverse phyla with aerobic and anaerobic thermophiles, anoxigenic phototrophs and anaerobes, able to respire organic halides, in the young Technosol complies with harsh edaphic conditions there. Although the relative abundance of Rhizobiales, Actinomycetales and unclassified Acidobacteria_Gp6 and Acidobacteria_Gp3 was found to be 1.5–2.4 times higher in the undisturbed Phaeozem, they also were predominating in the Technosol samples. The α-biodiversity indices, pertaining mostly to species/OTUs richness, i.e. Chao-1, OTUs number, Margalef, Fisher’s alpha, were higher in Phaeozem, whereas evenness and equitability were higher in Technosol. Both soils demonstrated similar Shanno indices, which were rather high (6.3).
Conclusions. Nine years of spontaneous revegetation on the terminated fly ash dump of the thermoelectric power station resulted in the development of the Technosol, containing quite rich and diverse bacterial assemblage, which was rather close in structure at the phylum taxonomic level to the adjacent undisturbed Phaeozem. Overall the early stages of pedogenesis were dominated by specific bacterial assemblage (Chloroflexi, Xanthomonadales, Geobacter, Aciditerrimonas, Iamiaceae and some others), performing weathering of the pedogenic substrate, i.e. fly ash. The present of nitrificators (Nitrospira), denitrificators денитрификаторов (Reyranella) together with diazotrophs (Bradyrhizobium, Rhizobiales) dominance evidences the establishment of the entire network of nitrogen transformation process already at the early stages of revegetation of the fly ash dump. Large percentage (17%) of Bacteria, the information about which ribosomal gene sequences is most likely absent in the respective data bases necessitates more detailed research into the soil microbiome on fly ash dumps.
Key words: Technosol; Phaeozem; fly ash dump; thermoelectric power station waste; bacterial assemblage; 16S rrNA genes; Illumina MiSeq; birch forest; West Siberia
How to cite: Naumova N.B., Belanov I.P., Alikina T.Y. Taxonomic diversity of bacterial assemblage in technosol of the revegetating fly ash dump // The Journal of Soil and Environment. 2019. 2(3). e84. 10.31251/pos.v2i3.84 (in Russian with English abstract).
References
- Androkhanov V.A., Kulyapina E.D., Kurachev V.M. Soil of technogenic landscapes: genesis and evolution. Novosibirsk: Nauka SB RAS Publ., 2004. 205 p. (in Russian)
- Kalachev A.I. A complex system of fly ash materials utilization, Consortium Fenix, June 2017, p.3 (in Russian)
- Soil classification and diagnostic of Russia / Authors and compilers: L.L. Shishov, V.D. Tonkonogov, I.I. Lebedeva, M.I. Gerasimov. Smolensk: Oykumena Publ., 2004. 342 p. (in Russian)
- The Climate of Novosibirsk and its changes / Luchitskaya I.O., Belaya N.. Arbuzov S.A. Federal Agency of Hydrometeorology and Environment Monitoring, Siberian Regional Hydrometeorological Research Institute. Novosibirsk: SB RAS Pubs., 201 224 p. (in Russian)
- Naumova N.B., Alikina T.Y., Kuznetsova G.V. Biodiversity of bacterial assemblages in the Haplic Cambisol under Korean pine, The Journal of Soils and Environment, 1(3), 151-169. CrossRef (in Russian)
- Shilov S.O., Shilova E.A. Selecting an option for fly ash dump extension on the basis of estimate calculation, The Synergy of sciences, 2017, No. 12, p. 821−827. Full text (in Russian)
- Energy industry and industry of Russia. 2019. March. Iss.5 (361). Full text (in Russian)
- Bayer-Santos E., Ceseti L.de M., Farah C.S., Alvarez-Martinez C.E. Distribution, Function and Regulation of Type 6 Secretion Systems of Xanthomonadales, Frontiers in Microbiology, 2019. CrossRef
- Berg J., Brandt K.K., Al-Soud W.A., Holm P.E., Hansen L.H., Sorensen S.J., Nybroe O. Selection for Cu-tolerant bacterial communities with altered composition, but unaltered richness, via long-term Cu exposure, Applied and Environmental Microbiology, 2012, V.78(20), p. 7438–7446. CrossRef
- Bergmann G. T., Bates S.T., Eilers K.G., Lauber C.L., Caporaso J.G., Walters W.A., Knight R., Fierer N.The under-recognized dominance of Verrucomicrobia in soil bacterial communities, Soil Biology and Biochemistry, 2011, 43(7), p. 1450–1455. CrossRef
- Brewer T., Handley K., Carini P. et al. Genome reduction in an abundant and ubiquitous soil bacterium ‘Candidatus Udaeobacter copiosus’, Nature Microbiology. 2017. V.2. art. 16198. CrossRef
- Castro-Silva C., Ruíz-Valdiviezo, V. M, Valenzuela-Encinas C. et al. The bacterial community structure in an alkaline saline soil spiked with anthracene, Electronic Journal of Biotechnology, 2013, 16(5). CrossRef
- Chen L, Feng Q, Li C. et al. Impacts of aquaculture wastewater irrigation on soil microbial functional diversity and community structure in arid regions, Scientific Reports, 2017а, V.7(1), Art.11193. CrossRef
- Chen X.W., Wong J.T., Leung A.O. et al. Comparison of plant and bacterial communities between a subtropical landfill topsoil 15years after restoration and a natural area, Waste Management, 2017b, 63(49-57). CrossRef
- Daims H., Lücker S., Wagner M. A New Perspective on Microbes Formerly Known as Nitrite-Oxidizing Bacteria, Trends in Microbioloogy. 2016, V. 24, p. 699–712. CrossRef
- Dukunde A., Schneider D., Schmidt M. Et al. Tree Species Shape Soil Bacterial Community Structure and Function in Temperate Deciduous Forests, Frontiers in microbiology, 2019, V.10, Art. 1519. CrossRef
- Edgar R.C. Search and clustering orders of magnitude faster than BLAST, Bioinformatics, 2010, 26(19), p. 2460-2461. CrossRef
- Edgar R.C. UPARSE: highly accurate OTU sequences from microbial amplicon reads, Nature Methods, 2013, V.10, p. 996–998. CrossRef
- Edgar R.C. UNOISE2: Improved error-correction for Illumina 16S and ITS amplicon reads, bioRxiv, 2016a. CrossRef
- Edgar R. C. SINTAX, a Simple Non-Bayesian Taxonomy Classifier for 16S and ITS Sequences, bioRxiv, 2016b. CrossRef
- Eichorst S.A., Kuske C.R. Identification of Cellulose-Responsive Bacterial and Fungal Communities in Geographically and Edaphically Different Soils by Using Stable Isotope Probing, Applied and Environmental Microbiology, 2012, 78(7), p. 2316–2327. CrossRef
- Fauth E., Bernardo J., Camara M. et al. Simplifying the Jargon of Community Ecology: A Conceptual Approach, The American Naturalist, 1996, V.147. P.282-286.
- Guo A., Ding L., Tang Z. et al. Microbial response to CaCO3 application in an acid soil in southern China, Journal of Environmental Science (China), 2019, V.79, p.321-329. CrossRef
- Hammer O., Harper D.A.T., Ryan P.D. PAST: Paleontological Statistics Software Package for Education and Data Analysis, Palaeontologia Electronica, 2001, 4(1), p.9.
- Haynes R.J. Reclamation and revegetation of fly ash disposal sites - Challenges and research needs, J Environ Manage, 2009, 90(1), p. 43-53. CrossRef
- Itoh T., Yamanoi K., Kudo T. et al. Aciditerrimonas ferrireducens gen. nov., sp. nov., an iron-reducing thermoacidophilic actinobacterium isolated from a solfataric field, International Journal of Systematic and Evolutionary Microbiology, 2011, 61(Pt 6), p.1281-1285. CrossRef
- IUSS Working Group, 2014. WRB, World Reference Base for Soil Resources 2015. International soil classification system for naming soils and creating legends for soil maps. FAO, Rome.203 p.
- Kasanke C.P., Collins R.E., Leigh M.B. Identification and Characterization of a Dominant Sulfolane-Degrading Rhodoferax sp. via Stable Isotope Probing Combined with Metagenomics, Scientific Reports, 2019, V.9. 3121. CrossRef
- Kim S.J., Ahn J.H., Lee T.H. et al. Reyranella soli sp. nov., isolated from forest soil, and emended description of the genus Reyranella Pagnier et al. 2011 // International Journal of Systematic and Evolutionary Microbiology. 2013. 63(Pt 9). P.3164-3167. CrossRef
- Kirillova I.V., Argant J., Lapteva E.G., Korona O.M., J. van der Plicht., Zinovyev E.V., Kotov A.A., Chernova O.F., Fadeeva E.O., Baturina O.A., Kabilov M.R., Shidlovskiy F.K., Zanina O.G. The diet and environment of mammoths in North-East Russia reconstructed from the contents of their feces, Quaternary International, 2016, V. 406, p. 147-161. CrossRef
- Krzmarzick M.J., Crary B.B., Harding J.J., Oyerinde O.O., Leri A.C., Myneni S.C., Novak P.J. Natural niche for organohalide-respiring Chloroflexi, Applied and Environmental Microbiology, 2012, 78(2), p. 393-401. CrossRef
- Lovley D.R., Walker D.J. F. Geobacter Protein Nanowires, Frontiers in Microbiology, 2019, V.10, p. 2078. CrossRef
- Lovley D.R., Ueki T., Zhang T. et al. Geobacter: the microbe electric's physiology, ecology, and practical applications, Advances in Microbial Physiology, 2011, V.59, p.1-100. CrossRef
- McCaig A.E., Glover L.A., Prosser J.I. Molecular analysis of bacterial community structure and diversity in unimproved and improved upland grassland pastures, Applied and Environmental Microbiology, 1999, 65(4), p. 1721-1730.
- Naumova N.B., Kuznetsova G.V., Alikina T.Y., Kabilov M.R. Bacterial 16S DNA diversity in the rhizosphere soil of two pine species, Biomics. 2015, 7(2), p.127-136. Full text
- Nunes I., Jacquiod S., Brejnrod A., etb al. Coping with copper: legacy effect of copper on potential activity of soil bacteria following a century of exposure, FEMS Microbiology Ecology, 2016, 92(11), fiw175. CrossRef
- Palomo A., Dechesne A., Smets B.F. Genomic profiling of Nitrospira species reveals ecological success of comammox Nitrospira, bioRxiv, 2019, 612226. CrossRef
- Pell M., Stenstrom L., Granhall U. Soil respiration, Microbiological methods for Assessing Soil Quality. Wallingford: CABI International, 2005, p.117-126.
- Pen-Mouratov S., Shukurov N., Yu J. Successive development of soil ecosystems at abandoned coal-ash landfills, Ecotoxicology, 2014, 23(5). p.880-897. CrossRef
- Ranjan K., Paula F.S., Mueller R.C. Forest-to-pasture conversion increases the diversity of the phylum Verrucomicrobia in Amazon rainforest soils, Frontiers in Microbiology, 2015, V.6, Art.779. CrossRef
- Sheremet N., Belanov I., Doronkin V., Lamanova T., Naumova N. Biogeocenosis development during initial revegetation of a coal combustion ash dump. In book: BIO Web of Conferences 2018. 11. 00038 Prospects of Development and Challenges of Modern Botany. CrossRef
- Soil Sampling and methods of Analysis. 2nd edition. Boca Raton: CRC Press; 2008. 1224 p.
- VanInsberghe D., Maas K. R., Cardenas E., Strachan C. R., Hallam S. J., Mohn W.W. Non-symbiotic Bradyrhizobium ecotypes dominate North American forest soils, The ISME journal, 2015, 9(11). p. 2435-2441. CrossRef
- Yamada T., Sekiguchi Y. Cultivation of Uncultured Chloroflexi Subphyla: Significance and Ecophysiology of Formerly Uncultured Chloroflexi 'Subphylum I' with Natural and Biotechnological Relevance, Microbes and environments. JSME, 2009, V.24, p. 205-216. CrossRef
Received 27 November 2019; accepted 19 December 2019; published 19 December 2019
About the authors:
Naumova Natalia – Candidate of Biological Sciences, Leading Researcher in the Laboratory of Agrochemistry in the Institute of Soil Science and Agrochemistry of the Siberian Branch of the Russian Academy of Sciences (Novosibirsk, Russia); naumova@issa-siberia.ru
Belanov Ivan – Candidate of Biological Sciences, Senior Researcher in the Laboratory of Soil Reclamation in the Institute of Soil Science and Agrochemistry of the Siberian Branch of the Russian Academy of Sciences (Novosibirsk, Russia); belanov@issa-siberia.ru
Аlikina Tatiana – Junior Researcher in the “Genomics” Core Facility, Institute of Chemical Biology and Fundamental Medicine, SB RAS, Novosibirsk, Russia; alikina@niboch.nsc.ru
The authors read and approved the final manuscript
 The article is available under Creative Commons Attribution 4.0 License
The article is available under Creative Commons Attribution 4.0 License
